绝对定量全转录组(mRNA、miRNA、lncRNA)
转录组测序利用高通量测序技术,快速且全面地揭示某一物种特定细胞或组织在某一状态下的几乎所有转录本信息,包括转录本表达量、可变剪接体、可变polyA位点等。
转录组测序结果准确性极大的受到文库扩增时PCR偏好性的影响:测序文库的构建过程无法避免使用PCR扩增。在PCR扩增时,所有的文库片段并非以同等速率等量的扩增,扩增速率受到片段长度、GC含量、片段浓度等多方面的影响,容易扩增的片段极大的被富集,一些含量较低的片段或碱基偏好严重的片段甚至完全丢失,最终影响测序结果的准确性。
绝对定量全转录组测序(KC-DigitalRNA-seq)利用数字标签(UMI)技术,追溯每一个文库片段,实现绝对化、数字化的精准定量。
建库原理
绝对定量转录组测序在文库扩增之前为每一条逆转录的cDNA片段加上唯一的身份标签(Unique Identifier,UMI),也称为数字标签。数字标签会伴随片段扩增、测序、分析的全过程。
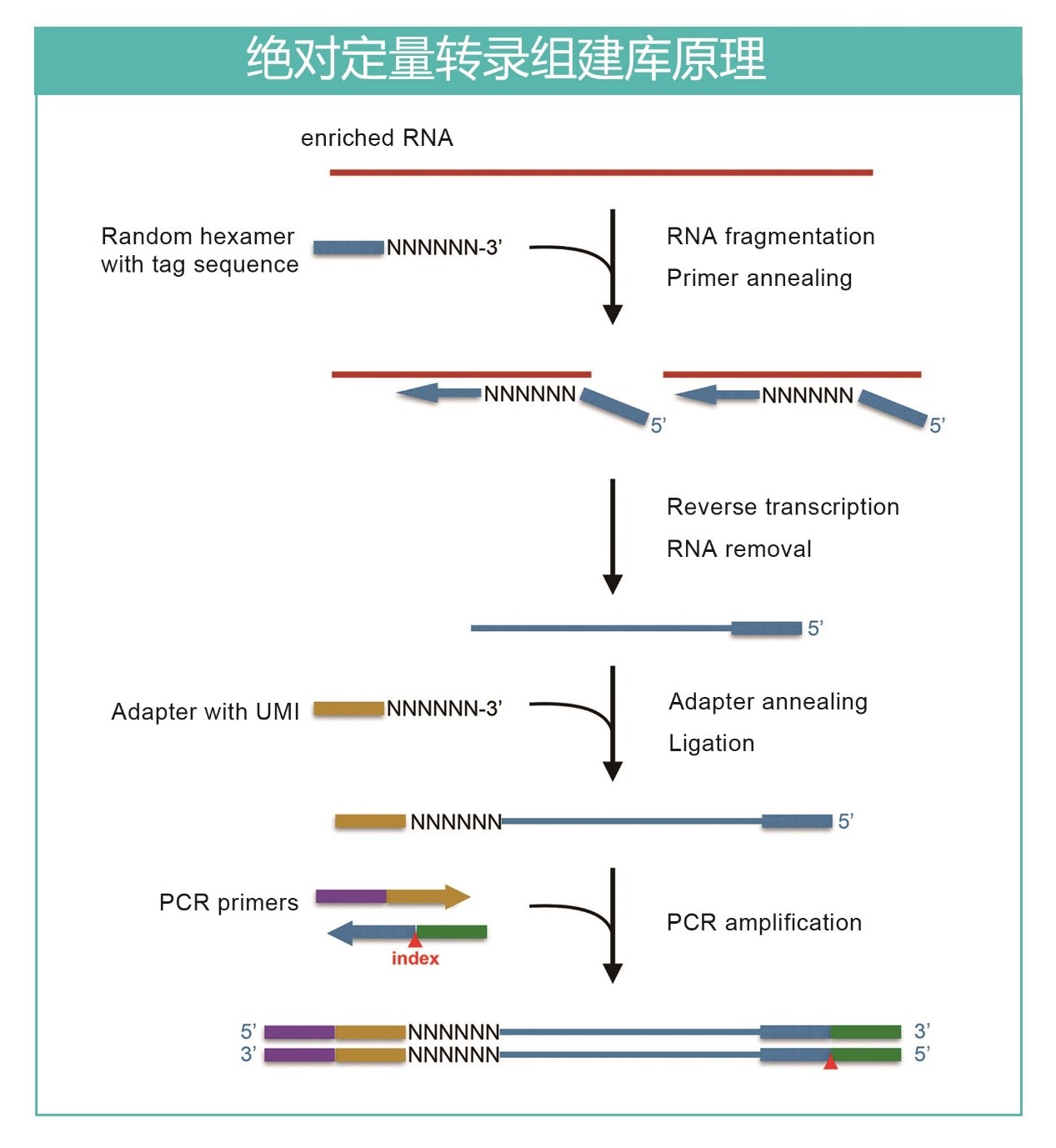
技术原理
建库完成之后,同一个片段经过PCR扩增出来的产物均带有相同的数字标签,测序完成后利用UMI追溯每一个片段的来源,将相同来源的片段(具有相同的序列和UMI)进行合并,就能准确去除PCR扩增重复,一比一准确还原样本扩增前的原始状态。在此过程中,PCR扩增和测序错误同样可以被纠正:扩增和测序的错误会使得相同UMI标签对应多个不同的序列,那么只需比较这些序列的相似性,即可纠正这些错误。
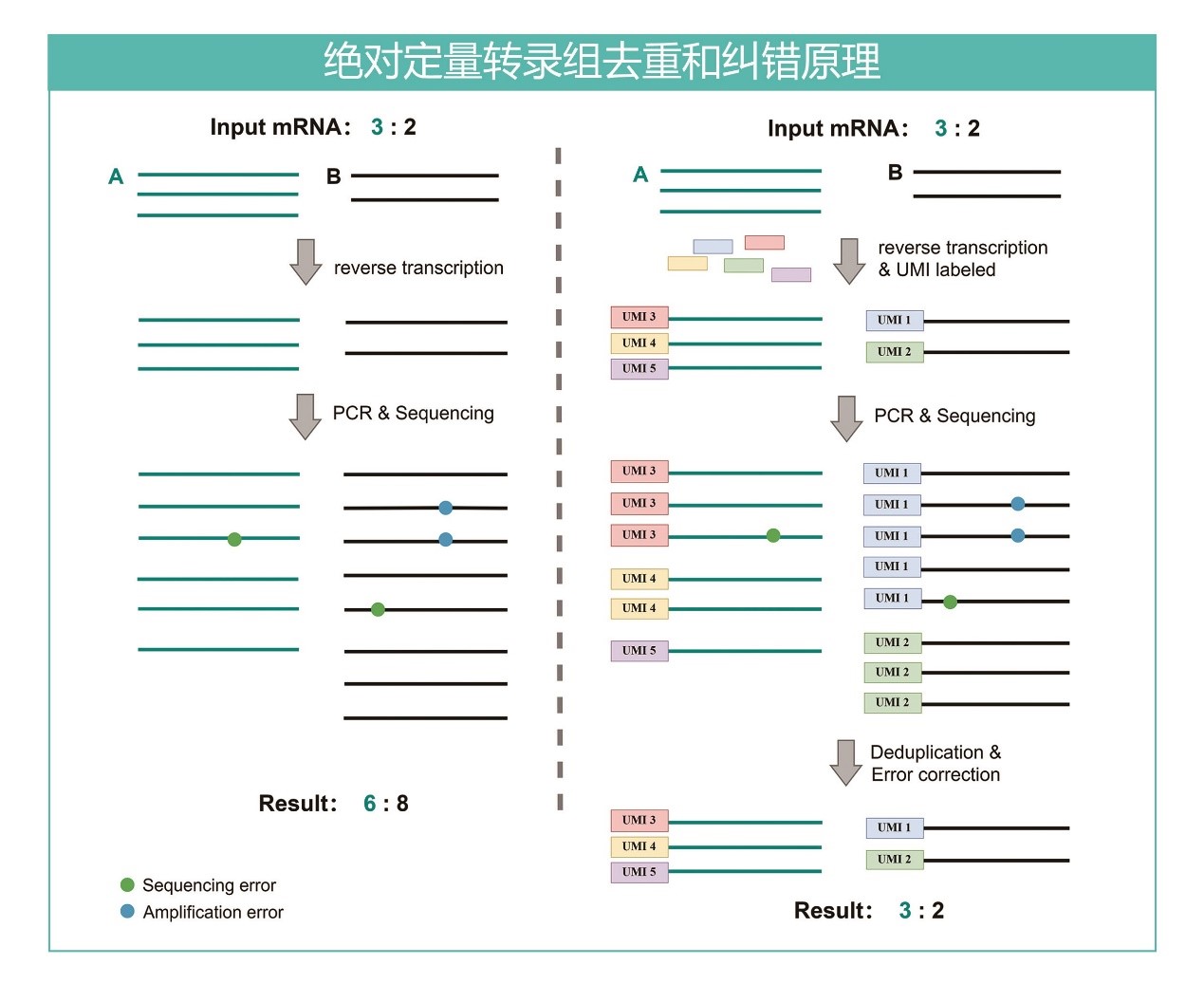
图 常规转录组测序(左)与绝对定量转录组测序(右)
技术优势
· 专利建库技术:为每一条RNA片段加上身份标签UMI,使测序全程可追溯;UMI标签去除PCR扩增偏好干扰,还原样本组成;
· 全面提升转录组测序质量:精确分析表达量和筛选差异表达,还能准确锁定RNA编辑、可变剪接、cSNP位点;
· 降低建库起始量:100ng即可达到1ug建库同等测序结果,更适合稀少珍贵的样本;
· 提高重复可靠性:排除PCR扩增偏好,多次重复可靠性高;
· 适用多种RNA测序:UMI建库及分析技术可适用mRNA、lncRNA、miRNA测序,以及RIP、CLIP、MeRIP等获得的RNA测序。
痛点解析
转录组测序数据(clean data)的重复序列由样本天然重复、PCR扩增重复、测序重复等组成,通常转录组测序中具有重复的测序序列(duplicated reads)占比20%~50%,无重复的序列(unique reads)仅占20%左右。通过UMI识别、去除PCR扩增和测序重复,可减少10~30%的重复序列,unique reads比例提高约10%。(左图,横坐标为reads的重复次数,纵坐标为此类reads占总reads的比例。)
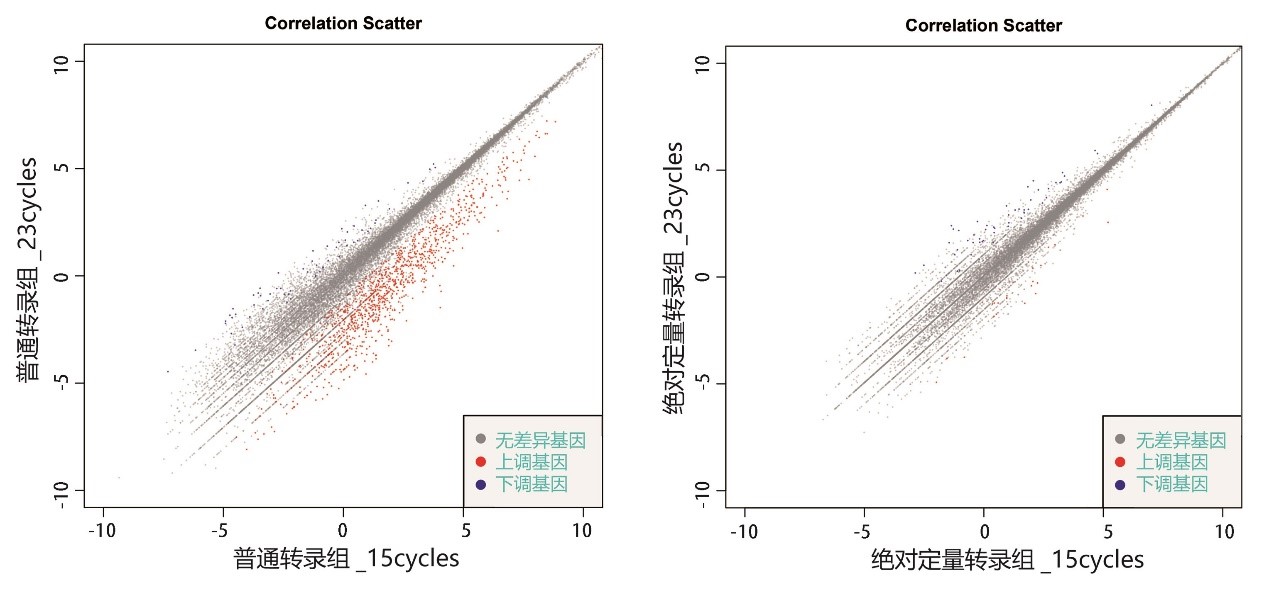
UMI可以消除PCR扩增偏好引入的结果差异
左图:使用普通RNA建库试剂盒对同一个样品在不同扩增条件下获得的基因表达的一致性; 右图:使用KC-digitalRNA建库试剂盒对同一个样品在不同扩增条件下获得的基因表达的一致性。
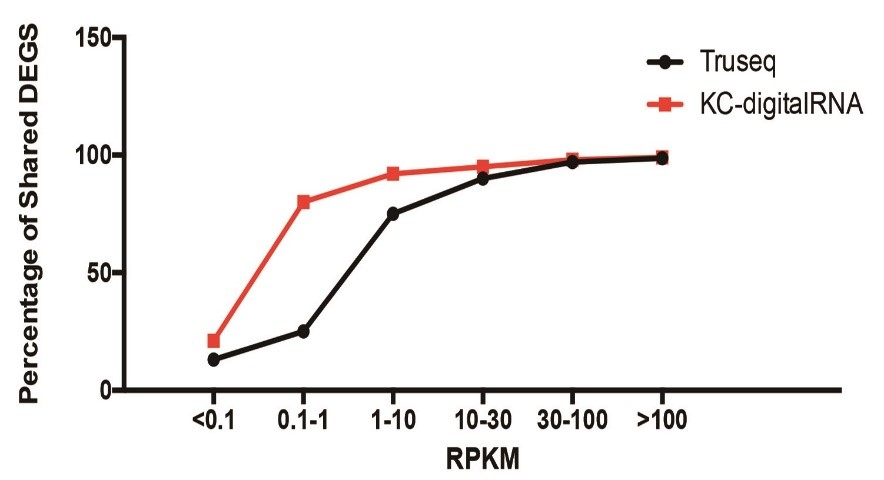
重复对不同表达水平基因DEGs(差异表达基因))鉴定的影响
通过对2个样品间的差异基因进行重复检测,左图为不同批次检测结果间一致性的展示。使用普通建库方法,表达量较低的基因,检测结果的重复性较差(黑色),使用KC-digitalRNA对重复进行处理后,中低表达量基因表达差异批次间检出的一致性有显著提高。
结果展示对比
对测序筛选的差异表达基因进行qPCR实验验证
选择48个差异表达基因(DigitalRNA-seq特有、RNA-seq特有及共有的差异表达基因各16个)进行qPCR验证,分析qPCR和测序结果的相关性。
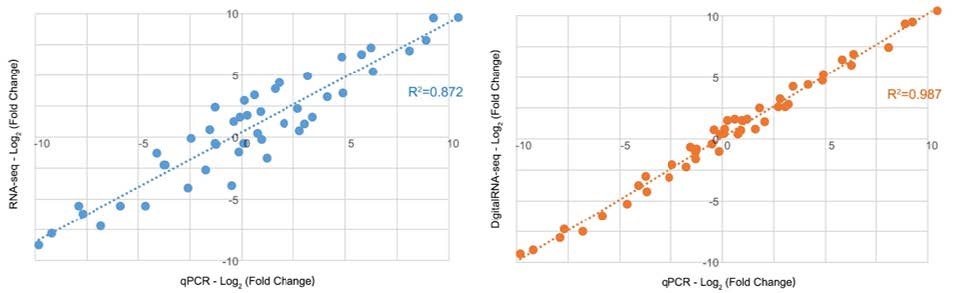
结果说明:
1. 左图为普通RNA-seq与qPCR实验结果的比较,右图为KC-DigitalRNA-seq与qPCR实验结果的比较;
2. 普通RNA-seq与qPCR实验结果的相关系数R2为0.872,KC-DigitalRNA-seq与qPCR实验结果的相关系数R2为0.987,可见KC-DigitalRNA-seq测序结果与qPCR结果非常相符,有很高的一致性。
KC-DigitalRNA-seq使用不同建库起始量的测序结果比较
分别使用100ng、500ng、1ug作为建库起始量,并将不同建库起始量的测序结果进行相关性分析。

结果说明:
100ng~1ug建库起始量的测序结果的Pearson相关系数关系数R2均在0.97以上。使用100ng起始建库,就能实现与1ug
高度一致的测序结果。